Mise à jour sur l’épidémiologie de la COVID-19 : Dépistage et variants:
- Dernière mise à jour : 2024-07-04
- Télécharger la page en format PDF
Résumé du dépistage et des variants de COVID-19 au Canada et au fil du temps. Des versions archivées de ce rapport sont disponibles.
La dernière mise à jour de cette page était le .
Modification du rapport
À compter du 4 octobre 2024, cette page web ne sera plus mise à jour. Les renseignements sur la COVID-19 sont désormais disponibles sur le rapport canadien de surveillance des virus respiratoires, où ils sont présenté avec les renseignements d'autres virus respiratoires, notamment la grippe et le VRS.
Sur cette page
Dépistage au Canada
Pour obtenir de l’information sur les virus respiratoires en circulation au Canada, consultez :
La présentation des données sur le dépistage de la COVID-19 ainsi que d’autres virus respiratoires, comme la grippe et le VRS, nous permet de prendre des décisions plus éclairées. Cela fait partie de notre travail en cours afin d’intégrer les rapports sur la COVID-19 et les virus respiratoires.
Variants au Canada
Tous les virus, y compris le SRAS-CoV-2 responsable de la COVID-19, changent au fil du temps. Ces changements sont appelés mutations, et les virus présentant des mutations sont appelés variants. On procède au séquençage du génome entier d’un certain pourcentage des échantillons porteurs du virus (résultat positif au test PCR de dépistage de la COVID-19) au Canada. Le séquençage nous indique le variant en cause dans un cas de COVID-19 particulier.
À propos des variants et des lignées
De nombreux variants font l’objet d’un suivi au Canada et dans le monde. Certains variants sont classés dans les catégories suivantes :
- Variant sous surveillance
- fait l’objet d’une surveillance afin d’évaluer ses mutations et ses caractéristiques
- Variant d’intérêt
- présente des mutations ou des caractéristiques d’intérêt et fait l’objet d’une surveillance pour déterminer s’il comporte un risque important pour la santé publique
- Variant préoccupant
- présente des mutations et des caractéristiques qui sont importantes pour la santé publique
Pour obtenir des définition illées, consultez Variants du SRAS-CoV-2 : Définitions, désignations et mesures de santé publique nationales.
Il arrive qu’une personne soit infectée par 2 variants différents en même temps. Les variants peuvent alors échanger des fractions de leur matériel génétique pour former un variant hybride, aussi appelé « virus recombinant ». Ces virus héritent des propriétés de chacun des virus parents, ce qui peut modifier le comportement du virus. Les variants mentionnés ci-dessous dont le nom scientifique commence par la lettre « X » sont des variants recombinants (par exemple, XBB.1).
Certains virus évoluent rapidement, entraînant l’émergence de nombreux variants au fil du temps. Pour simplifier le suivi, les variants sont regroupés en lignées (variants partageant un ancêtre commun récent). Par exemple, le variant BA.1 (sous-variant original d’Omicron) a créé plusieurs lignées telles que BA.1.1 et BA.1.1.1.
Depuis mars 2023, l’Organisation mondiale de la Santé (OMS) attribue des lettres grecques uniquement aux variants préoccupants. Les variants d’intérêt et les variants sous surveillance sont désignés à l’aide des systèmes de nomenclature scientifique en vigueur. Il n’y a actuellement aucun variant préoccupant en circulation au Canada, car Omicron a fait l’objet d’une « désescalade ».
Un variant fait l’objet d’une « désescalade » lorsqu’il apparaît clairement qu’il ne présente pas un risque élevé pour la population ou qu’il est remplacé par de nouveaux variants.
Variants récents
Le graphique montre la proportion des lignées de variants détectés au Canada par séquençage du génome entier au cours des 10 dernières semaines. Chaque semaine est représentée par une colonne. La lignée dominante pour chaque semaine est celle qui occupe la plus grande surface de la colonne.
Les 8 premières semaines de la figure reflètent les données réelles de la base de données génomiques nationale. Les 2 dernières semaines sont des estimations basées sur le modèle de prévision immédiate. La prévision immédiate utilise des modèles statistiques pour évaluer la situation actuelle en fonction des tendances antérieures. Elle fournit des estimations pour les dernières semaines lorsque les données sont encore recueillies et donc incomplètes.
Pour en savoir plus sur la prévision immédiate, consultez Méthodes de prévision immédiate.
Les valeurs numériques pour chaque lignée sont indiquées dans la légende et sont accompagnées d’intervalles de prévision. Un intervalle de prévision est une plage de valeurs (par exemple, de 16 à 19 %) qui indique le niveau de confiance de chaque estimation. Plus l’intervalle est petit, plus la certitude quant à la précision de la prévision est grande.
Figure 1. Ventilation par semaine des cas causés par des variants Mise à jour : mai 28, 2024, 9 h HE télécharger les données hebdomadaires de répartition des variants au format .csv
Pour voir les chiffres correspondant à une date, placez le curseur de la souris ou cliquez sur la colonne pertinente, ou appuyez sur la touche de tabulation. Pour obtenir les chiffres relatifs à un variant ou à un groupe de variants en particulier, cliquez ou appuyez sur Entrée. Cliquez à nouveau pour revenir au graphique complet. Les sous-variants peuvent être affichés ou masqués en cliquant sur le nom du variant dans la légende.
La collecte, le séquençage et le traitement des génomes viraux prennent du temps, il faut donc souvent prévoir 2 à 3 semaines pour le traitement des données. Nous utilisons un modèle de prévision immédiate pour estimer les proportions des variants pendant cette période.
* Englobe toutes les lignées descendantes, sauf indication contraire.
† Des données supplémentaires sont nécessaires pour estimer avec plus de certitude la croissance et la proportion de cette lignée.
Important : Lors de l’émergence d’une nouvelle lignée, les niveaux de détection sont trop faibles pour que la lignée soit incluse dans le modèle de prévision immédiate. Une fois la lignée prise en compte, les données demeurent limitées au départ, et la croissance et la proportion globale sont estimées de manière moins fiable. Au fur et à mesure que les données s’accumulent, l’exactitude et la précision des prévisions s’améliorent et l’intervalle de prévision diminue. Pendant les périodes de ralentissement de la collecte de données, les proportions globales peuvent être faussées et les prévisions, moins précises. Dans ces conditions, il convient d’interpréter les projections du modèle avec prudence.
Ventilation par semaine des cas causés par des variants
Tableau 1. Pourcentage des cas de COVID-19 cernés par séquençage du génome entier, présentés par variant et par semaine de collecte des échantillons
Figure 1: Texte descriptif
| Regroupements de variants | Variants |
|---|
Remarque : Les colonnes ombrées à droite représente une période d'accumulation de données.
Laboratoires d’origine
- Laboratoire national de microbiologie (LNM) — séquençage additionnel pour l’ensemble des provinces et des territoires
Séquençage des génomes du SARS-CoV-2 au Canada
Le Canada dispose d’un solide programme de séquençage génomique des virus. Les autorités de santé publique de tout le pays prélèvent et analysent des échantillons de tests basés sur la PCR pour identifier le variant présent dans chaque échantillon.
Les échantillons prélevés chez des personnes soupçonnées d’être atteintes de la COVID-19 sont analysés. Le matériel provenant des tests positifs est envoyé au laboratoire où le matériel génétique viral, ou acide ribonucléique (ARN), est extrait. Un échantillon est préparé et passé dans un appareil de séquençage. Celui-ci identifie les bases nucléotidiques présentes dans la séquence d’ARN. Il en résulte des chaînes de lettres qui sont assemblées pour donner le code génétique du variant de l’échantillon. Le code génétique du virus est utilisé pour classer et nommer le variant.
Les séquences virales nous indiquent également les variants qui sont présents au Canada, la façon dont ils se propagent et si les changements génétiques ont des répercussions sur la santé publique.
Figure 3. Façon dont le Canada séquence les génomes du SARS-CoV-2
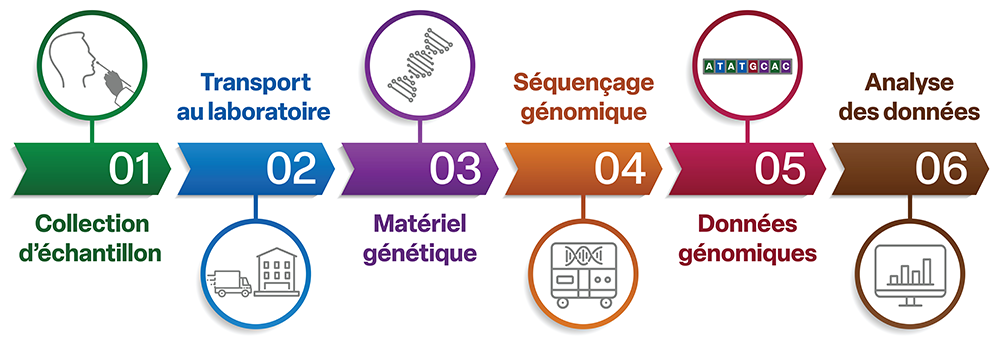
Figure 3: Texte descriptif
Le diagramme montre la manière dont le Canada procède au séquençage des génomes du SARS-CoV-2.
- Étape 1 : Collecte d’échantillon.
- Étape 2 : Transport au laboratoire.
- Étape 3 : Matériel génétique.
- Étape 4 : Séquençage génomique.
- Étape 5 : Données génomiques.
- Étape 6 : Analyse des données.
Données additionnelles sur la COVID-19
Ces produits pourraient aussi vous intéresser :
Tableau de bord sur la surveillance de la COVID-19 dans les eaux usées
Données sur les tendances relatives au niveau d’infection à la COVID-19 dans les eaux usées.
Vaccination contre la COVID-19
Nombre de doses de vaccin COVID-19 qui ont été administrées au Canada.
- Date de modification:
